Nathaniel L. Lartey , Salvador Valle-Reyes, Hilda Vargas-Robles, Karina E. Jiménez-Camacho, Idaira M. Guerrero-Fonseca, Ramón Castellanos-Martínez, Armando Montoya-García, Julio García-Cordero, Leticia Cedillo-Barrón, Porfirio Nava , Jessica G Filisola-Villaseñor, Daniela Roa-Velázquez, Dan I Zavala-Vargas, Edgar Morales-Rios, Citlaltepetl Salinas-Lara, Eduardo Vadillo and Michael Schnoor. ADAM17/MMP inhibition prevents neutrophilia and lung injury in a mouse model of COVID-19. Journal of Leukocyte Biology : 1-12: 2021.
doi: 10.1002/JLB.3COVA0421-195RR
Julio García-Cordero, Juvenal Mendoza-Ramírez, David Fernández-Benavides, Daniela Roa-Velázquez, Jessica Filisola-Villaseñor, Sandra Paola Martínez-Frías, Erik Saul Sánchez-Salguero, Carlos E. Miguel-Rodríguez, José L. Maravillas Montero, José J. Torres-Ruiz, Diana Gómez-Martín, Leopoldo Santos Argumedo, Edgar Morales-Rios, Juan M. Alvarado-Orozco and Leticia Cedillo-Barrón. Recombinant Protein Expression and Purification of N, S1, and RBD of SARS-CoV-2 from Mammalian Cells and Their Potential Applications. Diagnostics, MDPI 11: 1-14: 2021.
doi: 10.3390/diagnostics11101808
Leandro Núñez-Muñoz, Gabriel Marcelino-Pérez, Berenice Calderón-Pérez, Miriam Pérez-Saldívar, Karla Acosta-Virgen, Hugo González-Conchillos, Brenda Vargas-Hernández, Ana Olivares-Martínez, Roberto Ruiz-Medrano, Daniela Roa-Velázquez, Edgar Morales-Rios, Jorge Ramos-Flores, Gustavo Torres-Franco, Diana Peláez-González, Jorge Fernández-Hernández, Martha Espinosa-Cantellano, Diana Tapia-Sidas, José Abraham Ramírez-Pool, América Padilla-Viveros and Beatriz Xoconostle-Cázares. Recombinant Antigens Based on Non-Glycosylated Regions from RBD SARS-CoV-2 as Potential Vaccine Candidates against COVID-19. Vaccines, MDPI 9: 1-15: 2021.
doi: 10.3390/vaccines9080928
Ruben D. Arroyo-Olarte, Ricardo Bravo Rodríguez and Edgar Morales-Rios. Genome Editing in Bacteria: CRISPR-Cas and Beyond. Microorganisms, MDPI. 9: 1-25: 2021.
doi: 10.3390/microorganisms9040844
Urnavicius L, Lau CK, Elshenawy MM, Morales-Ríos E, Motz C, Yildiz A, Carter AP. Cryo-EM shows how dynactin recruits two dyneins for faster movement. Nature. (2018); Feb 7;554(7691):202-206;
doi: 10.1038/nature25462
Edgar Morales-Ríos, Martin G Montgomery, Andrew G W Leslie y John E Walker. The structure of ATP synthase from Paracoccus denitrificans determined by X-Ray crystallography at 4 Å resolution. PNAS. (2015);
Oct 27;112(43):13231-6;
doi: 10.1073/pnas.1517542112
Edgar Morales-Ríos, Ian N Watt, Qifeng Zhang, Shujing Ding, Ian M Fearnley, Martin G Montgomery, Michael JO Wakelam y John E Walker. Purification, characterisation and crystallization of the F-ATPase from Paracoccus denitrificans. Open Biology. (2015); Sep;5(9):150119;
doi: 10.1098/rsob.150119
Edgar Morales-Ríos, Martin G Montgomery, Andrew G W Leslie, José de Jesús García Trejo y John E Walker. Structure of a catalytic dimer of the a- and ß- subunits of the F-ATPase from Paracoccus denitrificans at 2.3 Å resolution. Acta crystallographica. (2015); Oct;71(Pt 10):1309-17;
doi: 10.1107/S2053230X15016076
Irma Eloisa Monroy-Muñoz, Nonanzit Pérez-Hernández, José Manuel Rodríguez-Pérez, José Esteban Muñoz-Medina, Javier Ángeles-Martínez, José J. García-Trejo, Edgar Morales-Ríos, Felipe Massó, Juan Pablo Sandoval-Jones, Jorge Cervantes-Salazar, José Antonio García-Montes, Juan Calderón-Colmenero y Gilberto Vargas-Alarcón. Transcriptional Activator Domain of the Human TBX20 in Patients with Atrial Septal Defect. (2015) BioMed Research International. 2015:718786;
doi: 10.1155/2015/718786
Mariel Zarco-Zavala, Edgar Morales-Ríos, Guillermo Mendoza-Hernández, Leticia Ramírez-Silva, Gerardo Pérez-Hernández y José de de Jesús García Trejo. Novel control mechanism of the F1FO-ATP synthase nanomotor of a-proteobacteria by the inhibitory ? subunit. The FASEB Journal. (2014); May;28(5)2146-57;
doi: 10.1096/fj.13-241430
Edgar Morales-Ríos, Fernanda de la Rosa-Morales, Guillermo Mendoza-Hernández, José S. Rodríguez-Zavala, Heliodoro Celis, Mariel Zarco-Zavala y José de Jesús García-Trejo. A novel 11-kDa inhibitory subunit in the F1FO ATP synthase of Paracoccus denitrificans and related a-proteobacteria. The FASEB Journal. (2010); Feb;24(2):599-608;
doi: 10.1096/fj.09-137356
José de Jesús García-Trejo y Edgar Morales-Ríos. Regulation of the F1FO-ATP Synthase Rotary Nanomotor in its Monomeric-Bacterial and Dimeric-Mitochondrial Forms. Journal of Biological Physics.(2008); Apr;34(1-2):197-212;
doi: 10.1007/s10867-008-9114-z
José de Jesús García-Trejo, Edgar Morales-Ríos, Paulina Cortés-Hernández y José Salud Rodríguez-Zavala. The inhibitor protein (IF1) promotes dimerization of the mitochondrial F1FO-ATP synthase. Biochemistry. (2006); Oct 24;45(42):12695-703;
doi: 10.1021/bi060339j
Concepción Bravo, Fernando Minauro-Sanmiguel, Edgar Morales-Ríos, José S. Rodríguez-Zavala y José J. García. Overexpression of the inhibitor protein IF1 in AS-30D hepatoma produces a higher association with mitochondrial F1FO ATP synthase compared to normal rat liver: functional and cross-linking studies. Journal of bioenergetics and biomembranes. (2004); Jun 36(3):257-64;
doi: 10.1023/B:JOBB.0000031977.99479.ea
Estudio estructural de motores moleculares y su relación con enfermedades degenerativas
Los motores moleculares son complejos multi-protéicos que producen importantes trabajos mecánicos o químicos ya sea utilizando adenosin trifosfato (ATP), el combustible energético de la célula, o produciéndolo.
El laboratorio se encarga de analizar a dos motores moleculares mediante estudios estructurales;
- La dineína humana es un complejo multi-proteíco que tiene un peso de 1,400 kDa y funciona como un motor molecular. Este motor se divide en dos dominios (Fig. 1); el “motor” que es el que utiliza ATP para “caminar” secuencialmente sobre los microtúbulos y; el dominio “tallo” donde se encuentra el dominio de dimerización y donde se unen las demás subunidades. La dineína está compuesta por dos copias de seis diferentes proteínas. Este motor molecular, junto con su cofactor, la dinactina, se encargan de la organización espacial, del transporte intra-celular en el sentido de la membrana hacia el núcleo y, además asisten en la división celular. Por otro lado, funciones de importancia médica, es que le dineína es reclutada para el transporte viral hacia el núcleo de la célula. Además, mutaciones de este complejo provocan severos trastornos neurodegenerativos en pacientes. En este proyecto, se purificarán fragmentos de la dineína humana original y mutantes para resolver sus estructuras cristalográficas a alta resolución y así proponer el mecanismo de este motor molecular con respecto a su unión con los diferentes cargos.
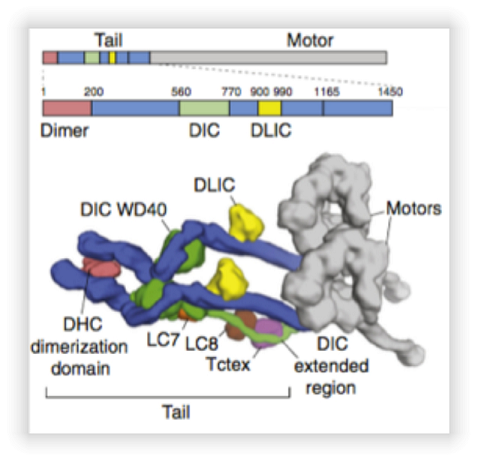
Figura 1. Modelo de la dineína humana derivado de crio-microscopía electrónica. Tomado de (Carter, AP et. al. 2016). En el panel superior se muestra la topología en escala de las diferentes subunidades de la dineína. El panel inferior muestra la localización aproximada de las subunidades y diferentes dominios de la dineína.
- La ATP sintasa es también un complejo protéico que se encuentra en las membranas transductoras de energía en membranas plasmáticas de bacteria, en las lamelas de cloroplasto de plantas y en la membrana interna de las mitocondrias de eucariontes, el peso molecular varía de 500 kDa a 850 kDa dependiendo del organismo en el que se encuentre. La ATP sintasa sintetiza ATP a partir de ADP y Pi usando la fuerza protón motriz como la fuente de energía. La enzima puede ser separada en dos sectores: El sector F1, el cual es hidrosoluble y es donde se alojan los sitios catalíticos; y el sector Fo hidrofóbico, embebido en la membrana, aquí, se encuentra el canal por el que pasan los protones que provoca el movimiento rotacional de la enzima. Gracias a los últimos estudios llevados a cabo en la ATP sintasa de Paracoccus denitrificans se ha logrado resolver una pregunta que se había mantenido en el campo por los últimos 30 años, la estructura cristalográfica de la enzima completa (Fig. 2). Con esta estructura se tiene un mejor entendimiento del funcionamiento del canal de protones y su acoplamiento a la síntesis de ATP. Una de las subunidades que había permanecido elusiva, la subunidad “a” yace en la membrana a 30o con respecto al eje central del anillo de subunidades “c” (Fig. 3). Mutaciones en esta subunidad a generan algunas enfermedades degenerativas. En este proyecto se resolverá la estructura atómica de la ATP sintasa con las diferentes mutaciones en la subunidad a para, en conjunto con técnicas bioquímicas, determinar el mecanismo molecular que provocan estas mutaciones en la enzima.

Figura 2. Cristalización y difracción de la ATP sintasa de Paracoccus denitrificans. (a) Cristales de la enzima, la escala mide 50 µm. (b) Gel de poli-acrilamida desnaturalizante en presencia de SDS de cristales disueltos. (c) Imagen de difracción de un cristal representativo.
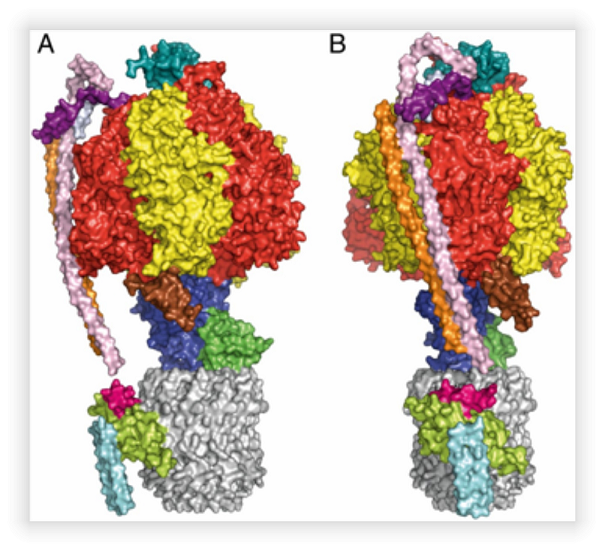
Figura 3. Modelo derivado de la difracción de los cristales de la ATP sintasa de P. denitrificans. A. Vista lateral del complejo. B. Vista frontal de la enzima.